AlphaFold2 讲解(2)
21年7月16日,Alphafold2和Rosettafold同时发表在Nature和Science上,并都在8月登上了Nature和Scinece的封面,自此,Protein folding成为了21年后半年所有人关注的焦点,Alphafold2可以干什么,后续高校和公司都在follow些什么内容,这一讲将详细捋清楚Alphafold2诞生之后的时间线,聊一聊Alphafold2能干什么,不能干什么,后续可以探究什么。
这篇文章缕一下脉络,详细的模型细节和模型设计的原因会在后续的文章中进一步来探究
在1972年的诺贝尔化学奖获奖感言中,克里斯蒂安·安芬森 (Christian Anfinsen ) 提出了一个 著名的假设 :理论上,蛋白质的 氨基酸序列 应该完全决定其结构。这一假设引发了 5 年的探索,即能够仅根据蛋白质的 1D 氨基酸序列计算预测蛋白质的 3D 结构,作为这些昂贵且耗时的实验方法的补充替代方案。
1994 年, John Moult 教授和 Krzysztof Fidelis 教授创立了 CASP 作为两年一次的盲测,以促进研究、监测进展并确立蛋白质结构预测的最新技术。它既是评估预测技术的黄金标准,也是建立在共同努力基础上的独特全球社区。
CASP 用来衡量预测准确性的主要指标是 全球距离测试 (GDT) ,范围为 0-100。简单来说, GDT 可以近似地认为是距正确位置的阈值距离内的氨基酸残基(蛋白质链中的珠子)的百分比。根据 Moult 教授 的说法,大约 90 GDT 的分数被非正式地认为与通过实验方法获得的结果具有竞争力。在CASP14结果中,Alphafold2在所有目标的总体得分为 92.4GDT ,意味着大约有1.6埃的平均误差(RMSD),与原子的宽度相当,在Free-modeling(无模板)也有 87.0GDT 。

结果让人震惊,下面是一些人对结果的评价
- DeepMind
https:// deepmind.com/blog/artic le/alphafold-a-solution-to-a-50-year-old-grand-challenge-in-biology
AlphaFold: a solution to a 50-year-old grand challenge in biology.
# Alphafold2解决了蛋白质折叠问题持续了50年的问题- PROFESSOR JOHN MCGEEHAN
https:// deepmind.com/research/c ase-studies/alphafold
约翰·麦吉汉教授 朴茨茅斯大学结构生物学教授和酶创新中心 (CEI) 中心主任
What took us months and years to do, AlphaFold was able to do in a weekend.
# 我们花费几月甚至几年的工作,Alphafold2可以在一个周末就做到- 小王笔记
https:// mp.weixin.qq.com/s? __biz=MzA4NjE2MzE1OA==&mid=2247485899&idx=1&sn=afc3dcf22431d547cc106d384a5ec3f3&chksm=9fcda272a8ba2b64e4fcd0a1561286432ecfd555eb12ec7744cdbf93b73984c51f1fa0184f9f&scene=178&cur_album_id=2094305991528873985#rd
AlphaFold2是一艘曲率飞船,从某种意义上说,在蛋白质科学领域,计算结果第一次拥有了与实验结果同等重要的地位,再也不能被轻视。毫无疑问,AlphaFold2对学术界和工业界产生了深远的影响,半年过去了,喧嚣过后,更需要我们去反思一下Alphafold2,现在我们可以做什么,挑战是什么,未来我们可以做什么。

21年7月15日Deepmind release 源码之后,敲响了工业界和学术界对于Alphafold2的探索和复现的路程。
截至到21年2月7日,目前Alphafold2的Citations已经有了 795 ,大家对Alphafold2做了各种有意思的研究和探索。

下面将基于探索和复现两个方面去阐述:
1. 探索
所有时间为论文首发的时间(发布在BioRxiv上的时间),而不是论文被接受的时间
- 21年7月16日 AlphaFold2 发布
Highly accurate protein structure prediction with AlphaFold | Nature
https:// github.com/deepmind/alp hafold
AlphaFold2的GDT为92.4;骨架碳原子RMSD为0.96埃,全原子RMSD为1.5埃;TM-score的成绩为90.3。
2. 21年7月16日RoseTTAFold 发布
Accurate prediction of protein structures and interactions using a three-track neural network (science.org)
https:// github.com/RosettaCommo ns/RoseTTAFold
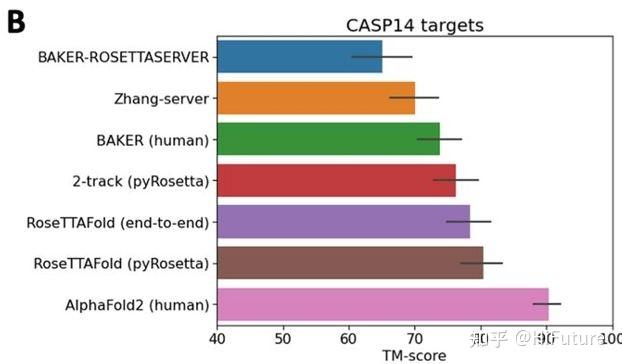
在CASP14中,AlphaFold2的TM-score的成绩为 90.3 ,RoseTTAFold的TM-score的成绩为 73.2
3. 21年7月17日 RoseTTAFold in google-colab
Sergey Ovchinnikov 发布colab版的RoseTTAFold,让使用者可以免于部署,在线运行
https:// colab.research.google.com /drive/1MBht6Nvy2vQCqPSdVg73zbiWZIvt7Zx0

4. 21年7月19日 AlphaFold2 in google-colab (民间版)
Sergey Ovchinnikov 发布colab版的AlphaFold2
旧版 https:// colab.research.google.com /drive/1LVPSOf4L502F21RWBmYJJYYLDlOU2NTL
新版 https:// colab.research.google.com /github/sokrypton/ColabFold/blob/main/AlphaFold2.ipynb
Sequence alignments/templates are generated through MMseqs2 and HHsearch
5. 21年7月23日 Deepmind realse AlphaFold2-colab (官方版)
https:// colab.research.google.com /github/deepmind/alphafold/blob/main/notebooks/AlphaFold.ipynb
与 AlphaFold v2.1.0 相比,此 Colab 笔记本 不使用模板(同源结构)和 BFD 数据库 的选定部分。我们已经在数千个最近的 PDB 结构上验证了这些更改。虽然在许多目标上准确度与完整的 AlphaFold 系统几乎相同,但由于较小的 MSA 和缺少模板,一小部分的准确度会大幅下降。
6. 21年7月19日 AlphaFold-Linker (G-link)
探索AlphaFold2预测多聚体的开端
AlphaFold2 还可以预测异质复合物。您所要做的就是输入您要预测的两个序列,并用一个长链接器将它们连接起来。
日本小哥 Yoshitaka Moriwaki
https:// twitter.com/Ag_smith/st atus/1417063635000598528

In this setup we add a 21 residue repeated Glycine-Glycine-Serine linker between each chain before running it as a single chain through the AlphaFold model. 【Glycine 甘氨酸,非必需氨基酸】
后续发现,U-link可能也会达到G-link类似的效果
https:// twitter.com/sokrypton/s tatus/1420218259798953989
https:// twitter.com/onoda_hirok i/status/1420117072839806977
显然,您可以使用聚“U”接头([U] 未知氨基酸)。获得与 G-link 相同的效果,但没有链接器妨碍。对于 U 链接器,建议 > 32(因为这是用于相对位置编码的最大 ij)
后续,就有文章利用G-link做peptide-protein docking
2021.8.1 Harnessing protein folding neural networks for peptide-protein docking
https://www. biorxiv.org/content/10. 1101/2021.08.01.454656v1

该文章参考:
Limits and potential of combined folding and docking using PconsDock. https://www. x-mol.com/paper/1402383 162367524864
21.7.27 Can AlphaFold2 predict protein-peptide complex structures accurately? https://www. biorxiv.org/content/bio rxiv/early/2021/07/28/2021.07.27.453972.full.pdf

8. 21年8月16日 AlphaFold-Gap(Colab-Fold)
ColabFold - Making protein folding accessible to all Milot
http:// github.com/sokrypton/Co labFold
9. 21年9月7日 结合Docking工具ClusPro预测聚合物
Improved Docking of Protein Models by a Combination of Alphafold2 and ClusPro
https://www. biorxiv.org/content/10. 1101/2021.09.07.459290v1.full.pdf
10 .21年9月30日 AlphaFold2 专利授权
Machine learning for determining protein structures
https:// patents.google.com/pate nt/US20210304847A1/en
11. 21年10月4日 Alphafold-Multimer发布
DeepMind发布AlphaFold-Multimer
https://www. biorxiv.org/content/10. 1101/2021.10.04.463034v1
12. 21年11月11日 结合RoseTTAFold和AlphaFold2大规模筛选真核生物PPI
Science文章
Computed structures of core eukaryotic protein complexes
https://www. science.org/doi/full/10 .1126/science.abm4805
13. AlphaDesign
21年10月1日 Michael Jendrusch
AlphaDesign: A de novo protein design framework based on AlphaFold
https://www. biorxiv.org/content/10. 1101/2021.10.11.463937v1.abstract
14. RFDesign
RFDesign: Protein hallucination and inpainting with RoseTTAFold
https:// github.com/RosettaCommo ns/RFDesign
2. 公司相关跟进
仅供参考,看看就好
1. BioMap百图生科
未发布具体细节和代码

# PR文:
# https://mp.weixin.qq.com/s/CZIJ4MlAzpplmjs1lJCmig
